SNP解析受託なら アメリエフ株式会社
2ケ月に一回行っている勉強会、そして今回は創立2周年記念パーティーに行ってきました。
私は過去2回、勉強会に参加しているのですが、勉強はもちろん、その後の懇親会でいろいろな方と話ができるのがとても有意義に感じています。
今回は2周年のパーティーということもあり、イタリアンレストランでの立食パーティーでした。
偶然にも、私の知り合い、お客さんも数人いました。 業界狭いですね。
アカデミック、企業、の垣根を越えたつながり。
同業だろうと気にせずに、みんなでバイオインフォマティクスを盛り上げよう!
という、社長の山口さんの考えに、私は大変共感します。
実際、会に参加しているひとの中には、私も含め企業の方もたくさんいます。
ライバル会社同士の方もいます。でも、そんなのは関係なし。
お酒が入ればざっくばらんな話ができます。
参加者は(こう言っては失礼ですが)お偉いさんは殆どおりません。
みな、20代から30代、40代の若手、いつも現場で汗かいている方々です。
変に気を使わなくても良いのがまたいいですね。
何人かの方が、私のこのブログをチェックされていました。
面等向かって言われると恥ずかしい・・・
ちゃんと書こう。
最後になりましたがこの場を借りて、アメリエフさん、2周年おめでとうございます!!
今後ともどうぞ宜しくお願いします。
ちょっと競合っぽくかぶるところもありますが、これからも飲み会、じゃなくって勉強会、誘ってください。
第2世代高速シーケンサー(Illumina, SOLiD, 454)とそのアプリケーションについて。 できるだけ最新に近いことと、トレンド、面白いと思った論文やアプリケーションを書いています。シーケンサーに興味のあるひとは是非覗いていってください。 コメントも大歓迎です。 一分子シーケンサー PacBioについては、姉妹ブログ「パックマンの挑戦 http://pacbiobrothers.blogspot.com/ 」 を、10X Genomicsについては「くろみうんの冒険」を覗いてみてください!
2011年6月25日土曜日
2011年6月24日金曜日
23andMe - 10万人を突破 遺伝子テストビジネスとその問題
Genetic Testing というサービスがあります。
数年前からアメリカやヨーロッパで流行り出し、サービス内容は、
という流れらしい。(上の絵は23andMeのサイトから)
らしい、と言ったのは、私はまだ実際にこのサービスを購入して試したことがないからです。
主なTesting会社は、23andMe、deCODEme、Navigenics, Pathway Genomics などがあります。
これらの会社は、今まではSNPアレイでジェノタイピングをしていましたが、今後、Complete Genomics社のような、パーソナルゲノムの時代になると、当然、アレイからゲノムへとシフトしていくのではないでしょうか?
これらGenetic Testingの会社が提供するサービスで、お客が得る情報は主に、
そのお客さんが先日10万人を突破したそうです。 記事はこちら
創業者の一人のAnne Wojcickiは、Google創業者の一人のSergey Brinの妻なのは有名です。
Googleがバックにあれば、将来が楽しみです。
一方、deCODE社やNavigenics社のように、専門家といつでもコンタクトして遺伝カウンセリングが受けられるサービスを売りにしている会社もあります。
この辺の歴史は、The $1,000 Genome: The Revolution in DNA Sequencing and the New Era of Personalized Medicineという本に詳しく書いてあります。 興味のある方はぜひ。


さて、遺伝子診断について、特定疾患に将来罹るリスク、というのは、実際いろいろな議論を呼んでいます。
2009年のNature に、"An agenda for personalized medicine" という記事があり、23andMeとNavigenicsの2社のサービスを5人が受けて結果を比較しています。
これらの会社は、今までのGWASなどから得られた様々な疾患リスクマーカーを元に、罹患者と健常人との比較で相対リスクを計算しています。
これに集団の平均罹患リスクとの掛け算をして、絶対リスクを出しています。
あなたの疾患Aに対する相対リスクが1.5で、疾患Aは集団の10%が罹患するものなら、あなたは1.5x10% = 15% の確率で将来罹患するでしょう。というものです。
先の記事によると、
同じ疾患マーカーを用いたケースでは、疾患のオッズ比(相対リスク)が2社の結果ともほぼ同じ(r=0.98)でした。 しかし、マーカーが異なる時はその限りではありません。
例えばpsoriasis (乾癬)について、被験者の一人は、23andMeで相対リスク4.02だったのに対し、Navigenicsでは1.25でした。3倍の差は大きいです。
これは使うマーカーの種類によるところが大きいのですが、会社では独自にマーカーの選別を行っており、それが3倍の差を生むのです。
また、集団の罹患リスクというものも、会社によって求め方に差があり、Navigenics社は性別を、23andMe社は年齢を、パラメータに入れて計算しているそうです。
ところでプラットフォームについても、NavigenicsはTaqManアレイ、23andMeはIlluminaアレイを使っていて、プローブやSNPの種類も異なります。
ひとつの会社でサービスを受けた時はどうでしょう。
マーカー選別のバイアスは無視して、結果を受け取り、Webで見たとします。
例えば、将来、心筋梗塞にかかる絶対リスクが60%あったとわかって、あなたはそれをどう受け取るでしょうか?
40%大丈夫と安心する?
60%もあるのかと心配する?
脂っこいものを控えて食生活を改善しようと思う?
軽い運動を始めてみる?
別の会社のテストを試してみる?
そして数ヵ月後、Webサイトが更新されて、そこには、「新しい疾患マーカーが発見されました!」 というニュースを見るのです。
「その新しい疾患マーカーを入れてリスクを再計算した結果、あなたの心筋梗塞リスクは30%に下がりました! おめでとうございます!」 と。
数年前からアメリカやヨーロッパで流行り出し、サービス内容は、
- お客はオンラインでキットを購入し
- 送られてきたキットを用いて、口腔内を特殊なへらでこすったり、チューブの中につばを入れ、
- キットを送り返す
- Genetic Testing会社は、マイクロアレイを使ってSNPジェノタイピングを行い、
- その結果を元に独自アルゴリズムで特定の疾患のリスクや、薬の副作用反応、先祖の系統、などを計算し、
- お客はWebで自分のデータを参照する
という流れらしい。(上の絵は23andMeのサイトから)
らしい、と言ったのは、私はまだ実際にこのサービスを購入して試したことがないからです。
主なTesting会社は、23andMe、deCODEme、Navigenics, Pathway Genomics などがあります。
これらの会社は、今まではSNPアレイでジェノタイピングをしていましたが、今後、Complete Genomics社のような、パーソナルゲノムの時代になると、当然、アレイからゲノムへとシフトしていくのではないでしょうか?
これらGenetic Testingの会社が提供するサービスで、お客が得る情報は主に、
- 遺伝的な祖先
- 特定疾患(癌や糖尿病など)に将来罹るリスク
- 薬(抗がん剤など)に対する副作用のリスク
- マーカー遺伝子、Mutationのあるなし
- これらの情報をソーシャルネットワークを使って他人と共有(23andMeのビジネスモデル)
- あるいは遺伝専門家のカウンセリング(deCODEme, Navigenicsのビジネスモデル)
そのお客さんが先日10万人を突破したそうです。 記事はこちら
創業者の一人のAnne Wojcickiは、Google創業者の一人のSergey Brinの妻なのは有名です。
Googleがバックにあれば、将来が楽しみです。
一方、deCODE社やNavigenics社のように、専門家といつでもコンタクトして遺伝カウンセリングが受けられるサービスを売りにしている会社もあります。
この辺の歴史は、The $1,000 Genome: The Revolution in DNA Sequencing and the New Era of Personalized Medicineという本に詳しく書いてあります。 興味のある方はぜひ。

さて、遺伝子診断について、特定疾患に将来罹るリスク、というのは、実際いろいろな議論を呼んでいます。
2009年のNature に、"An agenda for personalized medicine" という記事があり、23andMeとNavigenicsの2社のサービスを5人が受けて結果を比較しています。
これらの会社は、今までのGWASなどから得られた様々な疾患リスクマーカーを元に、罹患者と健常人との比較で相対リスクを計算しています。
これに集団の平均罹患リスクとの掛け算をして、絶対リスクを出しています。
あなたの疾患Aに対する相対リスクが1.5で、疾患Aは集団の10%が罹患するものなら、あなたは1.5x10% = 15% の確率で将来罹患するでしょう。というものです。
先の記事によると、
同じ疾患マーカーを用いたケースでは、疾患のオッズ比(相対リスク)が2社の結果ともほぼ同じ(r=0.98)でした。 しかし、マーカーが異なる時はその限りではありません。
例えばpsoriasis (乾癬)について、被験者の一人は、23andMeで相対リスク4.02だったのに対し、Navigenicsでは1.25でした。3倍の差は大きいです。
これは使うマーカーの種類によるところが大きいのですが、会社では独自にマーカーの選別を行っており、それが3倍の差を生むのです。
また、集団の罹患リスクというものも、会社によって求め方に差があり、Navigenics社は性別を、23andMe社は年齢を、パラメータに入れて計算しているそうです。
ところでプラットフォームについても、NavigenicsはTaqManアレイ、23andMeはIlluminaアレイを使っていて、プローブやSNPの種類も異なります。
ひとつの会社でサービスを受けた時はどうでしょう。
マーカー選別のバイアスは無視して、結果を受け取り、Webで見たとします。
例えば、将来、心筋梗塞にかかる絶対リスクが60%あったとわかって、あなたはそれをどう受け取るでしょうか?
40%大丈夫と安心する?
60%もあるのかと心配する?
脂っこいものを控えて食生活を改善しようと思う?
軽い運動を始めてみる?
別の会社のテストを試してみる?
そして数ヵ月後、Webサイトが更新されて、そこには、「新しい疾患マーカーが発見されました!」 というニュースを見るのです。
「その新しい疾患マーカーを入れてリスクを再計算した結果、あなたの心筋梗塞リスクは30%に下がりました! おめでとうございます!」 と。
2011年6月19日日曜日
Ion Torrent のデータセット
ヨーロッパで先月末に発生して1500人以上が感染、17人が死亡した、大腸菌O104-H4。
日本のニュースでも話題になりましたが、これは最初、新種の大腸菌ではないかと報道されました。
(読売新聞6月3日「欧州で広がるO104、新種の可能性…WHO」)
やがて、ドイツと中国、その他のチームによってゲノムが読まれ、これが新種ではないものの、ハイブリッドな特徴を持つ厄介な菌であることがわかりました。 BGIによると、
このE.coliは、下痢原性大腸菌の一つのカテゴリーである腸管凝集性大腸菌 enteroaggregative E. coli (EAEC) の系統ですが、志賀毒素を産生するファージゲノムを自身のバクテリアゲノムに組み込み、さらに多薬剤耐性遺伝子をもゲノムに組み込んでいたことがわかりました。
環状ゲノムのサイズは 5,278 kbp で、ほかに 88 kbp, 75 kbp, 1.5 kbp の3つのPlasmidから成るとのこと。
通常、志賀毒素を出す遺伝子、多剤耐性遺伝子は、ファージが関係するのですが、このE.coliは自身のゲノムにこれら遺伝子を組み込んでいることから、暫定的にShiga toxin-producing enteroaggregative Escherichia coli (STpEAEC) と呼ばれているそうです。
腸管出血性大腸菌 enterohaemorrhagic E. coli (EHEC) に特徴は似ているけれど別の名前で呼ばれているんですね。知りませんでした。
さて、前置きが長くなりました。
このドラフトシーケンシングに用いられた機器が、半導体シーケンサー Ion Torrent PGM です。
(実はBGIは、PGMのほかにIlluminaのマシンも使って読んでいるのですがそれはさておき)
Ion Torrent社のホームページへ行くと大きく宣伝しているのがわかるでしょう。
今はまだ数少ない、PGMのデータ(この大腸菌のリード)はここから入手できます。
Sffフォーマット: http://lifetech-it.hosted.jivesoftware.com/docs/DOC-1621
Fastqフォーマット: http://lifetech-it.hosted.jivesoftware.com/docs/DOC-1516
もしかするとユーザー登録が必要かもしれません。
また、アプリケーションノートやビデオなどは、Ion Torrentを良く知るために大変役に立ちますのでお勧めです。
念のため申しますと、私は Ion Torrent 社及びライフテクノロジーズ社と、特別な関係、があるわけではありませんのでご安心下さい。
ひとつ断わりをいれておきますが、Ion Torrent社のサイトから落とせるデータは、BGIが読んだデータではありません。
ストレインが違います。
現在5つのストレインでゲノムが読まれていて
フォーマットはfastqです。
解析、de novo assembly については、彼ら(BGI)のワークフローが参考になるかと思います。
バクテリアゲノムアセンブリを初めてされる方は見ておいて損は無いでしょう。
http://climb.genomics.cn/Ecoli_TY-2482
見てわかる通り、BGIのチームは、IlluminaとIon Torrentの両方で読んで、アセンブルしました。
Ion Torrent PGM データのアセンブリに、Newbler を使ったのですね。
Roche 454 と同じ sff フォーマットなのでこれが使えるのでしょう。
PGMは、sff を出した後、fastqでも出力してくれます。 これならvelvetなどフリーツールでも使えますね。
現在、PGM に付属するソフトには、残念ながら独自のアセンブリツールが無いので、今のところPGMを使って de novo assembly をしようとすると、アセンブリソフトを別に求める必要があります。
さて、Ion Torrent のサイトから入手できる、LB226692のデータに話を戻しますね。
先のサイトから、sff のファイルをダウンロードしてきました。
8ファイル(8ラン分)あります。
私はRocheの機械を持っていませんし、アカデミアではなく企業の人間なので、タダでNewblerは使えません。
fastqを落としてアセンブってもいいがせっかく sff のフォーマットがあるのでできればsffのままアセンブりたい。
さて、、、どのアセンブリツールを使おうか。
手元にちょうどCLC Genomics Workbench があるのでそれでやってみることにします。
最初に言い訳がましくなりますが、このPGMのデータだけを使ってアセンブリしても、1本にはなりません。
BGIは、7ランのPGMのデータ、200x以上のIlluminaのシングルリード、さらにIlluminaペアエンド、を使って読んでやっとドラフトゲノムを完成させています。
私がこれからお見せしようとしているのは、PGMのsffデータだけでアセンブルしたら、どれくらいのContigができるのか、というものです。
CLC のGenomics Workbenchには、sff フォーマットがインポートでき、アセンブルも簡単にできます。
取り込んだ後の様子
アセンブルした後のマッピングした結果のContigリスト
Contigをクリックしたときの様子
データ量: 8ラン分のデータ、平均98bpの117万リード、合計114Mbp
アセンブルに要した時間: 1分10秒
マシンの性能: 64Bit Linux, メモリ24Mb
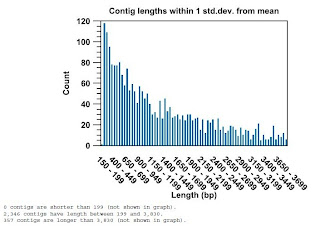
Contigについて(200bp以上の長さのみ)
2,700本のContigができました。 最長16,000 bpでした。
ふーん、こんなもんかな。
こんどはちゃんと配列の前処理をやってからランしようかと、思った次第です。
と、ここで終わってしまっては結構さみしいので、次に、BGIのTY2482のリードデータを使って、
Ion Torrent 7ラン分 + Illumina 200x分 のハイブリッドアセンブリをやってみました。
フォーマットはfastq です。 結果をまとめると、
データ量: 合計 20.5 M reads, 1.8 G bp
ランタイム: 15分
Contigについて
512本のContig、最長224,799 bp、N50 = 60,618 bp!
実はBGIはペアエンドでも読んでいるのですが、データは公開されていません。
ゆえにこれは、Ion Torrent PGM +Illumina GAII シングリリード のハイブリッドアセンブリの結果、です。
ショートリード(GAII)とロングリード(PGM)がうまく互いを補いながら、長くつないでいるのでしょうね。
ちなみにCLC Genomics Workbench のパラメータはデフォルトです。
このソフトについてはこちらを参照
http://www.w-fusion.com/J/CLC_wb.html
・・・・・・今回は少し長文になりました。
大腸菌についての参考資料
広島県保健環境センター研究報告,No.12,p1-12,2004
http://www.pref.hiroshima.lg.jp/hec/press/pdf/kenkyuhoukoku12/01.pdf
日本のニュースでも話題になりましたが、これは最初、新種の大腸菌ではないかと報道されました。
(読売新聞6月3日「欧州で広がるO104、新種の可能性…WHO」)
やがて、ドイツと中国、その他のチームによってゲノムが読まれ、これが新種ではないものの、ハイブリッドな特徴を持つ厄介な菌であることがわかりました。 BGIによると、
このE.coliは、下痢原性大腸菌の一つのカテゴリーである腸管凝集性大腸菌 enteroaggregative E. coli (EAEC) の系統ですが、志賀毒素を産生するファージゲノムを自身のバクテリアゲノムに組み込み、さらに多薬剤耐性遺伝子をもゲノムに組み込んでいたことがわかりました。
環状ゲノムのサイズは 5,278 kbp で、ほかに 88 kbp, 75 kbp, 1.5 kbp の3つのPlasmidから成るとのこと。
通常、志賀毒素を出す遺伝子、多剤耐性遺伝子は、ファージが関係するのですが、このE.coliは自身のゲノムにこれら遺伝子を組み込んでいることから、暫定的にShiga toxin-producing enteroaggregative Escherichia coli (STpEAEC) と呼ばれているそうです。
腸管出血性大腸菌 enterohaemorrhagic E. coli (EHEC) に特徴は似ているけれど別の名前で呼ばれているんですね。知りませんでした。
さて、前置きが長くなりました。
このドラフトシーケンシングに用いられた機器が、半導体シーケンサー Ion Torrent PGM です。
(実はBGIは、PGMのほかにIlluminaのマシンも使って読んでいるのですがそれはさておき)
Ion Torrent社のホームページへ行くと大きく宣伝しているのがわかるでしょう。
今はまだ数少ない、PGMのデータ(この大腸菌のリード)はここから入手できます。
Sffフォーマット: http://lifetech-it.hosted.jivesoftware.com/docs/DOC-1621
Fastqフォーマット: http://lifetech-it.hosted.jivesoftware.com/docs/DOC-1516
もしかするとユーザー登録が必要かもしれません。
また、アプリケーションノートやビデオなどは、Ion Torrentを良く知るために大変役に立ちますのでお勧めです。
念のため申しますと、私は Ion Torrent 社及びライフテクノロジーズ社と、特別な関係、があるわけではありませんのでご安心下さい。
ひとつ断わりをいれておきますが、Ion Torrent社のサイトから落とせるデータは、BGIが読んだデータではありません。
ストレインが違います。
現在5つのストレインでゲノムが読まれていて
- TY2482 (BGI in collaboration with University Medical Centre Hamburg-Eppendorf)
- LB226692 (Life Tech in-house in collaboration with University of Muenster)
- H112180280 (Health Protection Agency, Colindale, UK)
- 2 isolates, unnamed (Gottingen Genomics Lab, Germany)
BGIはTY2482を読んでいます。
Ion Torrentのサイトから落とせるデータは、LB226692のデータです。
BGIのデータを使いたい方は、
NCBI SRA からは、SRX067313 で検索すると出てきます。
BGIのサイトからは、ftp://ftp.genomics.org.cn/pub/Ecoli_TY-2482フォーマットはfastqです。
解析、de novo assembly については、彼ら(BGI)のワークフローが参考になるかと思います。
バクテリアゲノムアセンブリを初めてされる方は見ておいて損は無いでしょう。
http://climb.genomics.cn/Ecoli_TY-2482
見てわかる通り、BGIのチームは、IlluminaとIon Torrentの両方で読んで、アセンブルしました。
Ion Torrent PGM データのアセンブリに、Newbler を使ったのですね。
Roche 454 と同じ sff フォーマットなのでこれが使えるのでしょう。
PGMは、sff を出した後、fastqでも出力してくれます。 これならvelvetなどフリーツールでも使えますね。
現在、PGM に付属するソフトには、残念ながら独自のアセンブリツールが無いので、今のところPGMを使って de novo assembly をしようとすると、アセンブリソフトを別に求める必要があります。
さて、Ion Torrent のサイトから入手できる、LB226692のデータに話を戻しますね。
先のサイトから、sff のファイルをダウンロードしてきました。
8ファイル(8ラン分)あります。
私はRocheの機械を持っていませんし、アカデミアではなく企業の人間なので、タダでNewblerは使えません。
fastqを落としてアセンブってもいいがせっかく sff のフォーマットがあるのでできればsffのままアセンブりたい。
さて、、、どのアセンブリツールを使おうか。
手元にちょうどCLC Genomics Workbench があるのでそれでやってみることにします。
最初に言い訳がましくなりますが、このPGMのデータだけを使ってアセンブリしても、1本にはなりません。
BGIは、7ランのPGMのデータ、200x以上のIlluminaのシングルリード、さらにIlluminaペアエンド、を使って読んでやっとドラフトゲノムを完成させています。
私がこれからお見せしようとしているのは、PGMのsffデータだけでアセンブルしたら、どれくらいのContigができるのか、というものです。
CLC のGenomics Workbenchには、sff フォーマットがインポートでき、アセンブルも簡単にできます。
取り込んだ後の様子
アセンブルした後のマッピングした結果のContigリスト
Contigをクリックしたときの様子
データ量: 8ラン分のデータ、平均98bpの117万リード、合計114Mbp
アセンブルに要した時間: 1分10秒
マシンの性能: 64Bit Linux, メモリ24Mb
Contigについて(200bp以上の長さのみ)
2,700本のContigができました。 最長16,000 bpでした。
ふーん、こんなもんかな。
こんどはちゃんと配列の前処理をやってからランしようかと、思った次第です。
と、ここで終わってしまっては結構さみしいので、次に、BGIのTY2482のリードデータを使って、
Ion Torrent 7ラン分 + Illumina 200x分 のハイブリッドアセンブリをやってみました。
フォーマットはfastq です。 結果をまとめると、
データ量: 合計 20.5 M reads, 1.8 G bp
ランタイム: 15分
Contigについて
512本のContig、最長224,799 bp、N50 = 60,618 bp!
実はBGIはペアエンドでも読んでいるのですが、データは公開されていません。
ゆえにこれは、Ion Torrent PGM +Illumina GAII シングリリード のハイブリッドアセンブリの結果、です。
ショートリード(GAII)とロングリード(PGM)がうまく互いを補いながら、長くつないでいるのでしょうね。
ちなみにCLC Genomics Workbench のパラメータはデフォルトです。
このソフトについてはこちらを参照
http://www.w-fusion.com/J/CLC_wb.html
・・・・・・今回は少し長文になりました。
大腸菌についての参考資料
広島県保健環境センター研究報告,No.12,p1-12,2004
http://www.pref.hiroshima.lg.jp/hec/press/pdf/kenkyuhoukoku12/01.pdf
2011年6月15日水曜日
PacBioのシーケンサーがあるのはどこだ?
こんなサイトをご存じでしょうか?
今、世界中で、どこの研究所にどんなシーケンサーが導入されているのかが、ひと目でわかるマップです。http://pathogenomics.bham.ac.uk/hts/
必ずしも全てを網羅しているわけではないと思いますが、どこにどんなシーケンサーが導入されているのか、(実際は今現在導入されているもの以外にも、導入がほぼ決定しているものも、含まれているようですが)がわかって面白いですね。
アジアではやはり中国のBGIが圧倒的! 137台のHiSeqですか・・・ 見てみたいです。137台のHiSeqが並んでいるその光景を。 でもRoche454が1台というのは意外です。 もっとあるんじゃないかな?
では今話題の、というか私が一番気にしている、PacBio RSがあるのはどこでしょう?
上の、PacBioのチェックボックスにチェックを入れると表示されます。
右側に、研究所の名前のリストも出てきますよ・・・
さすが北米には12台も!
ちなみに導入先(予定も含む)研究所の名前を見てみると、
・・・ Japanの名前は、まだ無いですね。
今年中には、PacBio のマシン&データに触れる機会がありそうですので、今から楽しみです。
今、世界中で、どこの研究所にどんなシーケンサーが導入されているのかが、ひと目でわかるマップです。http://pathogenomics.bham.ac.uk/hts/
必ずしも全てを網羅しているわけではないと思いますが、どこにどんなシーケンサーが導入されているのか、(実際は今現在導入されているもの以外にも、導入がほぼ決定しているものも、含まれているようですが)がわかって面白いですね。
アジアではやはり中国のBGIが圧倒的! 137台のHiSeqですか・・・ 見てみたいです。137台のHiSeqが並んでいるその光景を。 でもRoche454が1台というのは意外です。 もっとあるんじゃないかな?
では今話題の、というか私が一番気にしている、PacBio RSがあるのはどこでしょう?
上の、PacBioのチェックボックスにチェックを入れると表示されます。
右側に、研究所の名前のリストも出てきますよ・・・
さすが北米には12台も!
ちなみに導入先(予定も含む)研究所の名前を見てみると、
- NCGR Genome Center: NM, United States
- DOE Joint Genome Institute: CA, United States
- National Cancer Institute: MD, United States
- Wellcome Trust Sanger Institute: Cambridgeshire, United Kingdom
- Monsanto: MO, United States
- Tri-I biotech, inc.: Taipei County, Taiwan
- Institute for Genome Sciences, University of Maryland: MD, United States
- Ontario Institute for Cancer Research: ON, Canada
- Leiden University Medical Centre: Zuid-Holland, Netherlands
- Broad Institute: MA, United States
- Cold Spring Harbor Laboratory: NY, United States
- The Genome Center at Washington University: St. Louis, MO, United States
- Center for Genomics and Personalized Medicine Department of Genetics Stanford University School of Medicine: CA, United States
- Expression Analysis: NC, United States
- University of Michigan DNA Sequencing Core/Affymetrix and Microarray Core Facility: MI, United States
・・・ Japanの名前は、まだ無いですね。
今年中には、PacBio のマシン&データに触れる機会がありそうですので、今から楽しみです。
2011年6月14日火曜日
FPKM、RPKM
FPKMとRPKM、似ているようだが一体何なのか。
TopHat-Cufflinks の発現解析パイプラインでは、発現量をFPKMという方法で表していました。
もともとこれと似たようなものに、RPKMがあります。
FPKMは、Fragments Per Kilobase of exon per Million mapped fragments の略、
RPKMは、Reads Per Kilobase of exon per Million mapped reads の略です。
FragmentsかReadsかの違いだけですね。 言わんとすることは殆ど同じです。
つまり、ノーマライズした発現量 です。
もうちょっと詳しく言うと、例えば、
SAMPLE1とSAMPLE2、という、別のレーンでランをしたサンプルがあるとします。
Sample1では、リファレンス全体にマップされたリードが1000万本、Sample2では2000万本、ありました。
ここでGeneAに注目すると、Sample1では200本、Sample2では400本のReadが、GeneAにマップされていました。
リファレンス全体にマップされたリードの本数を無視すると、GeneAの発現量は、Sample1:Sample2 = 200 : 400 = 1 : 2 となりますが、これは全体の発現量でノーマライズするべきですね。
マイクロアレイでも、各プローブのシグナルを、全プローブのシグナルの中央値で割って、ノーマライズ(Median Normalize)することがありますが、あれと同じような考え方です。
リファレンス全体にマップされたReadの本数が、仮に100万本だったとき(i.e. 基準を100万本に揃えてノーマライズ)、各々の遺伝子にマップされたReadは何本に相当するか、と。
これが100万本でノーマライズしたときの、RPM (read per million) です。
さらに、RNA-Seqの場合、各々の転写産物の長さも考慮に入れなければいけません。
長い転写産物はそれだけ多くのReadがマップされるからです。
転写産物の長さは遺伝子によって様々、一方Readの長さは50 baseなら50 baseで一定ですから、長い遺伝子が多くマップされるのは当たり前ですね。
これも仮に、全ての転写産物が1,000 base 長としたとき、マップされたRead本数はいくつに相当するか、を算出することができます。
これが、転写産物1000塩基長でノーマライズしたときの、RPKM (read per kilobase / million)です。
異なる遺伝子間の発現量を比較するときは必須です。
RPKMはReadを単位にしていました。シングルリードで読んだ時はこちら。
FPKMはFragment、ペアエンドで読んだときの2つのReadを1組としたfragment、を単位としています。
尤も、RPKMやFPKMなどでノーマライズしたとしても、Read/fragment のカウンティングの方法によって、発現量の多い転写産物は実際よりもずっと多くカウントされてしまう、などのノイズ・バイアスがかかってくることもありますので、万能ではありません。
最近はそれらノイズを補正するアルゴリズムも公開されていますが、これらについてはまた。
(私も他人に説明できるレベルまで理解しきれていないので、こう言って逃げることをお許し下さい)
RPKMについて: Mortazavi et al. (2008). Mapping and quantifying mammalian transcriptomes by rna-seq. Nat Methods, 5(7):621--628.
FPKMについてはCufflinks等のサイト参照
TopHat-Cufflinks の発現解析パイプラインでは、発現量をFPKMという方法で表していました。
もともとこれと似たようなものに、RPKMがあります。
FPKMは、Fragments Per Kilobase of exon per Million mapped fragments の略、
RPKMは、Reads Per Kilobase of exon per Million mapped reads の略です。
FragmentsかReadsかの違いだけですね。 言わんとすることは殆ど同じです。
つまり、ノーマライズした発現量 です。
もうちょっと詳しく言うと、例えば、
SAMPLE1とSAMPLE2、という、別のレーンでランをしたサンプルがあるとします。
Sample1では、リファレンス全体にマップされたリードが1000万本、Sample2では2000万本、ありました。
ここでGeneAに注目すると、Sample1では200本、Sample2では400本のReadが、GeneAにマップされていました。
リファレンス全体にマップされたリードの本数を無視すると、GeneAの発現量は、Sample1:Sample2 = 200 : 400 = 1 : 2 となりますが、これは全体の発現量でノーマライズするべきですね。
マイクロアレイでも、各プローブのシグナルを、全プローブのシグナルの中央値で割って、ノーマライズ(Median Normalize)することがありますが、あれと同じような考え方です。
リファレンス全体にマップされたReadの本数が、仮に100万本だったとき(i.e. 基準を100万本に揃えてノーマライズ)、各々の遺伝子にマップされたReadは何本に相当するか、と。
これが100万本でノーマライズしたときの、RPM (read per million) です。
さらに、RNA-Seqの場合、各々の転写産物の長さも考慮に入れなければいけません。
長い転写産物はそれだけ多くのReadがマップされるからです。
転写産物の長さは遺伝子によって様々、一方Readの長さは50 baseなら50 baseで一定ですから、長い遺伝子が多くマップされるのは当たり前ですね。
これも仮に、全ての転写産物が1,000 base 長としたとき、マップされたRead本数はいくつに相当するか、を算出することができます。
これが、転写産物1000塩基長でノーマライズしたときの、RPKM (read per kilobase / million)です。
異なる遺伝子間の発現量を比較するときは必須です。
RPKMはReadを単位にしていました。シングルリードで読んだ時はこちら。
FPKMはFragment、ペアエンドで読んだときの2つのReadを1組としたfragment、を単位としています。
尤も、RPKMやFPKMなどでノーマライズしたとしても、Read/fragment のカウンティングの方法によって、発現量の多い転写産物は実際よりもずっと多くカウントされてしまう、などのノイズ・バイアスがかかってくることもありますので、万能ではありません。
最近はそれらノイズを補正するアルゴリズムも公開されていますが、これらについてはまた。
(私も他人に説明できるレベルまで理解しきれていないので、こう言って逃げることをお許し下さい)
RPKMについて: Mortazavi et al. (2008). Mapping and quantifying mammalian transcriptomes by rna-seq. Nat Methods, 5(7):621--628.
FPKMについてはCufflinks等のサイト参照
2011年6月3日金曜日
発現解析パイプラインを作るぞ! その2: TopHat の使い方 2
さて、無事TopHatが終了し、下のような画面が出て、
accepted_hits.bam, junctions.bed, insertions.bed / deletions.bed というファイルができたとしましょう。
最後のアウトプットに.bam と書くと、出力が.bam.bam と2重拡張子になるので注意!
次に、後でUCSCとかIGVとかのGenome Viewerで見るために、ソートしたBAMファイルにインデックスをつけておきます。
samtools index ./tophat_SRP003186_Control/sort_accepted_hits.bam
samtools index ./tophat_SRP003186_MCF7/sort_accepted_hits.bam
samtools index ./tophat_SRP003186_SKBR3/sort_accepted_hits.bam
拡張子.bai というファイルができるはずです。 これがインデックスです。
samtools view ./tophat_SRP003186_MCF7/sort_accepted_hits.bam -o ./tophat_SRP003186_MCF7/MCF7_sort_accepted_hits.sam
samtools view ./tophat_SRP003186_SKBR3/sort_accepted_hits.bam -o ./tophat_SRP003186_SKBR3/SKBR3_sort_accepted_hits.sam
1) FPKM tracking ファイル: トランスクリプトごとのFPKMを出力し、各cuffdiffランごとに4ファイルできる
isoforms.fpkm_tracking: Transcript FPKMs
genes.fpkm_tracking: Gene FPKMs. 同じgene_idを持つトランスクリプトのFPKMの積算
(cds.fpkm_tracking: Coding sequence FPKMs.)
(tss_groups.fpkm_tracking: Primary transcript FPKMs)
gene_exp.diff: Gene differential FPKM.
(tss_group_exp.diff: Primary transcript differential FPKM)
(cds_exp.diff: Coding sequence differential FPKM)
accepted_hits.bam, junctions.bed, insertions.bed / deletions.bed というファイルができたとしましょう。
この3ファイルが何かについては前回説明しました。
TopHatは、コマンド実行時に指定したgtfファイルを使って、gtfに書かれている既知の転写配列情報から、Exon Junction を認識してマッピングしてくれるのです。
Exon Junctionを認識するということは、Fusion Geneも特定してくれそうです。
それは、Tophat-fusion というツールがあるのですが、また今度別のときに書きます。
さて、うまくマッピングされ、BAMファイルができました。
では次に、Samtools を使って、BAMファイルを染色体番号順にソートします。
これは後で必要になります。
samtools sort ./tophat_SRP003186_Control/accepted_hits.bam ./tophat_SRP003186_Control/sort_accepted_hits
samtools sort ./tophat_SRP003186_MCF7/accepted_hits.bam ./tophat_SRP003186_MCF7/sort_accepted_hits
samtools sort ./tophat_SRP003186_SKBR3/accepted_hits.bam ./tophat_SRP003186_SKBR3/sort_accepted_hits最後のアウトプットに.bam と書くと、出力が.bam.bam と2重拡張子になるので注意!
samtools index ./tophat_SRP003186_Control/sort_accepted_hits.bam
samtools index ./tophat_SRP003186_MCF7/sort_accepted_hits.bam
samtools index ./tophat_SRP003186_SKBR3/sort_accepted_hits.bam
拡張子.bai というファイルができるはずです。 これがインデックスです。
ついでにソートしたBAMファイルを、SAMフォーマットにしておきましょうか? 別に必要無いですけど、samtools に慣れていないひとは練習がてら。
ちなみにIGVは、SAMファイルも取り込めます。その際には、IGV-toolsでインデックスをつけるひつようがあります。
また、以前紹介した、SAM-Mate というWindows 対応のRNA-SEQ解析ツールは、SAMでないと受け付けてくれません、今のところ。
samtools view ./tophat_SRP003186_Control/sort_accepted_hits.bam -o ./tophat_SRP003186_Control/Control_sort_accepted_hits.samsamtools view ./tophat_SRP003186_MCF7/sort_accepted_hits.bam -o ./tophat_SRP003186_MCF7/MCF7_sort_accepted_hits.sam
samtools view ./tophat_SRP003186_SKBR3/sort_accepted_hits.bam -o ./tophat_SRP003186_SKBR3/SKBR3_sort_accepted_hits.sam
発現変動を見るには、Cufflinksのコマンドのひとつ、Cuffdiff というものを実行します。
ソートしたBAMファイル、既知トランスクリプトのGTFファイルの2つを使います。
cuffdiff -p 4 -v /Path_to_gtf/refGene.gtf ./tophat_SRP003186_Control/sort_accepted_hits.bam ./tophat_SRP003186_MCF7/sort_accepted_hits.bam -o ./cuffdiff/MCF7
cuffdiff -p 4 -v /Path_to_gtf/refGene.gtf ./tophat_SRP003186_Control/sort_accepted_hits.bam ./tophat_SRP003186_SKBR3/sort_accepted_hits.bam -o ./cuffdiff/SKBR3コントロール v.s. MCF-7、コントロール v.s. SK BR-3 の2つの比較ですね。
本当はリプリケートがあると良かったのですが、これはN=1です。
パラメータは単純にデフォルトで行っています。
唯一、CUPを -p 4 にしたことくらいでしょうか。
Cuffdiffのアウトプットファイルは地味で、タブ区切りテキストファイルができるだけです。
もちろん加工すればGenome Viewer上でも表示できますが。
FPKM tracking ファイルと、Differential expression 結果ファイルの2種類があります。
isoforms.fpkm_tracking: Transcript FPKMs
genes.fpkm_tracking: Gene FPKMs. 同じgene_idを持つトランスクリプトのFPKMの積算
(cds.fpkm_tracking: Coding sequence FPKMs.)
(tss_groups.fpkm_tracking: Primary transcript FPKMs)
2) Differential expression 結果ファイル: 各cuffdiffランごとに4ファイル
isoform_exp.diff: Transcript differential FPKM. gene_exp.diff: Gene differential FPKM.
(tss_group_exp.diff: Primary transcript differential FPKM)
(cds_exp.diff: Coding sequence differential FPKM)
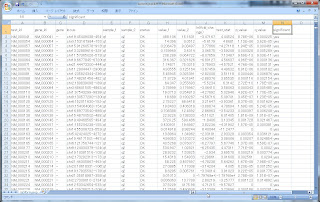
このうち、発現変動の結果ファイル、isoform_exp.diff をExcelで開いてみるとこんな感じです。
トランスクリプトのIDや名前、ポジション情報が先ずあり、そのあとHカラムくらいから、実際のカウンティングデータがきます。
- FPKMx: FPKM of the gene in sample x
- FPKMy: FPKM of the gene in sample y
- ln(FPKMy/FPKMx):The natural log (自然対数) of the fold change y/x
- test stat: FPKMの変動が有意であるかどうかの統計量
- p value: 補正無しの p-value
- q value: FDR-adjusted p-value
- significant:"yes" or "no" マルチサンプル比較の場合はBenjamini-Hochberg補正後、FDR-adjusted p-valueが、cuffdiffランのパラメータ --FDR (デフォルト:0.05)より小さい場合は、"Yes" 有意だとされる
Ratioは、自然対数のログ表記という点にご注意ください。
また、Excelはsignificant = Yes で絞り込んでいます。
こんな風にして、RNA-Seq の実験データから、発現変動の有意な遺伝子を解析することができるのです。
実際は、パラメータの調整などが必要ですが、感じはわかって頂けたと思います。
このあと考えられる操作は、
- 発現変動の有意だった遺伝子のポジション情報からBedファイルを作り、
- 先ほどソート済みのBAMファイルをGenome Viewerと一緒にとりこみ、
- ゲノム上で発現の変動と、マッピングの様子を同時に見る
ことでしょうか。 これはまた、Viewerに関する書き込みのときに詳しく説明します。
つづく
2011年6月2日木曜日
発現解析パイプラインを作るぞ! その1: TopHat の使い方
TopHatといえば、NGS発現解析で良く使われるツールです。
論文やポスターでも頻繁に見かけるので、一応、スタンダードなツールと見て良いでしょう。
TopHatと一緒にCufflinksという名前も、聞いたことがあるのではないでしょうか?
こちら、セットで使うと、RNA-Seqの解析にそこそこ威力を発揮します。
TopHatを使うには、Bowtie(高速マッピングツール、http://bowtie-bio.sourceforge.net/index.shtml)
と、Samtools (SAM<->BAMファイルの変換やSNP検出などをするツール、http://samtools.sourceforge.net/)も一緒にインストールしておくと便利です。
Bowtie、TopHat、Samtoolsは、インストールも比較的難なくできますが、Cufflinksはちょっと準備が必要(bjamとかいうエンジンを入れる必要)ですので、行き詰ったらLinuxに詳しい方に助けを乞いましょう。
4つともうまく入ったら、どんな風に動くのか、早速試してみましょう。
デモデータは、SRAから落としてきたSRP003186のデータを使います。 乳がんサンプル、イルミナGAIIのペアエンドデータです。
そうそう、SRAのファイルは、そのままでは解凍できないので、SRAのサイトからSRA-TOOLSというものを落としてきて、Linuxマシンにインストールします。 詳しくは昨年12月の書き込み(サンプルデータの取得法 2 NCBI 、http://shortreadbrothers.blogspot.com/2010/12/ncbi.html)にあるが、ここでのコマンドはfastq-dump2 から現在 fastq-dump に変わっているので注意!
全体の流れを示します。既知トランスクリプトーム情報を使った、発現変動解析パイプラインの大まかな例です。 必要以外のジョブもありますが、そこはつっこまないで。
24本の染色体だったら、24個のファイルができるので、それを cat *.fa > hg19.fa としてひとつのファイルにまとめる。
ついでに、UCSCから、好きなアノテーションファイル(GTFフォーマット)を落としてきます。
これについては、この画面を参照
インデックスは、Bowtieのbuildコマンドで
bowtie-build /Path_to_reference/UCSC_hg19/hg19.fa ./bowtie_ref/hg19
いよいよTopHatの実行です。
先ほどのgtfファイルを用意して、以下のようなコマンドを流します。 3つのサンプルがあります。
コマンドは3行です。ペアエンドなので、それぞれ***_1.fastq ***_2.fastq というファイル名です。
tophat -r 100 -p 4 -G /Path_to_gtf/refGene.gtf -o ./tophat_SRP003186_Control /Path_to_bowtie_ref/hg19 /Path_to_read_file/Control/SRR064437_1.fastq /Path_to_read_file/Control/SRR064437_2.fastq
tophat -r 0 -p 4 -G /Path_to_gtf/refGene.gtf -o ./tophat_SRP003186_MCF7/Path_to_bowtie_ref/hg19 /Path_to_read_file/MCF_7/SRR064286_1.fastq /Path_to_read_file/MCF_7/SRR064286_2.fastq
tophat -r 100 -p 4 -G /Path_to_gtf/refGene.gtf -o ./tophat_SRP003186_SKBR3 /Path_to_bowtie_ref/hg19 /Path_to_read_file/SK_BR_3/SRR064441_1.fastq /Path_to_read_file/SK_BR_3/SRR064441_2.fastq
アウトプットファイルは、たくさんできるのですが、以下の3ファイルがとくに重要!というか、後はあまり見ません。
つづく
論文やポスターでも頻繁に見かけるので、一応、スタンダードなツールと見て良いでしょう。
TopHatと一緒にCufflinksという名前も、聞いたことがあるのではないでしょうか?
こちら、セットで使うと、RNA-Seqの解析にそこそこ威力を発揮します。
ちなみにこの写真は、エイブラハム・リンカーンが、実際に使っていたTopHatです。
スミソニアンのアメリカ史博物館での一枚です。
残念ながらCufflinks(カフスボタン)は、飾っていませんでした。
冗談はさておき、TopHatは、Exon-Intronのジャンクションを挟んでうまくアライメントするマッピングツールです。 Cufflinksは、そのマップされたフラグメント(リード)をカウントするツールです。
TopHatはこちら(http://tophat.cbcb.umd.edu/index.html)
Cufflinksはこちらから(http://cufflinks.cbcb.umd.edu/)
ダウンロードできます。 マニュアルもあります。 Cufflinksは最近頻繁にバージョンアップされています。TopHatを使うには、Bowtie(高速マッピングツール、http://bowtie-bio.sourceforge.net/index.shtml)
と、Samtools (SAM<->BAMファイルの変換やSNP検出などをするツール、http://samtools.sourceforge.net/)も一緒にインストールしておくと便利です。
Bowtie、TopHat、Samtoolsは、インストールも比較的難なくできますが、Cufflinksはちょっと準備が必要(bjamとかいうエンジンを入れる必要)ですので、行き詰ったらLinuxに詳しい方に助けを乞いましょう。
4つともうまく入ったら、どんな風に動くのか、早速試してみましょう。
デモデータは、SRAから落としてきたSRP003186のデータを使います。 乳がんサンプル、イルミナGAIIのペアエンドデータです。
そうそう、SRAのファイルは、そのままでは解凍できないので、SRAのサイトからSRA-TOOLSというものを落としてきて、Linuxマシンにインストールします。 詳しくは昨年12月の書き込み(サンプルデータの取得法 2 NCBI 、http://shortreadbrothers.blogspot.com/2010/12/ncbi.html)にあるが、ここでのコマンドはfastq-dump2 から現在 fastq-dump に変わっているので注意!
全体の流れを示します。既知トランスクリプトーム情報を使った、発現変動解析パイプラインの大まかな例です。 必要以外のジョブもありますが、そこはつっこまないで。
- リファレンス配列Hg19を用意する
- Hg19に対してBowtieのインデックスを貼る
- TopHatを実行、リファレンスにマッピングする
- できたBAMファイルをソートしておく
- 後でViewerで見やすいようにソート後のBAMファイルにインデックスを貼る
- ついでにソートしたBAMファイルをSAMファイルにもしておく
- そのまたついでにソートしたBAMファイルをもとに、リファレンス配列と比較して異なる塩基(変異)のポジションを見つける
- さっき4でソートしたBAMファイルから、既知トランスクリプトのGTFファイルをもとに発現比較する ― Cuffdiff 実行
24本の染色体だったら、24個のファイルができるので、それを cat *.fa > hg19.fa としてひとつのファイルにまとめる。
ついでに、UCSCから、好きなアノテーションファイル(GTFフォーマット)を落としてきます。
これについては、この画面を参照
bowtie-build /Path_to_reference/UCSC_hg19/hg19.fa ./bowtie_ref/hg19
いよいよTopHatの実行です。
先ほどのgtfファイルを用意して、以下のようなコマンドを流します。 3つのサンプルがあります。
コマンドは3行です。ペアエンドなので、それぞれ***_1.fastq ***_2.fastq というファイル名です。
tophat -r 100 -p 4 -G /Path_to_gtf/refGene.gtf -o ./tophat_SRP003186_Control /Path_to_bowtie_ref/hg19 /Path_to_read_file/Control/SRR064437_1.fastq /Path_to_read_file/Control/SRR064437_2.fastq
tophat -r 0 -p 4 -G /Path_to_gtf/refGene.gtf -o ./tophat_SRP003186_MCF7/Path_to_bowtie_ref/hg19 /Path_to_read_file/MCF_7/SRR064286_1.fastq /Path_to_read_file/MCF_7/SRR064286_2.fastq
tophat -r 100 -p 4 -G /Path_to_gtf/refGene.gtf -o ./tophat_SRP003186_SKBR3 /Path_to_bowtie_ref/hg19 /Path_to_read_file/SK_BR_3/SRR064441_1.fastq /Path_to_read_file/SK_BR_3/SRR064441_2.fastq
パラメータについて
-r: inner mate distance 平均フラグメントの長さ-両側のリードの長さ。MCFの例の場合、フラグメント長は100塩基でリード長は50塩基なので、100-50x2=0-p: 使用するCUP数(デフォルト:1)
-o: アウトプットファイルを書き出すディレクトリ
-G: gtfファイルの場所
このほかのパラメータについてはおいおい。 もちろんTopHatのウェブサイトにも情報はたくさんあります。アウトプットファイルは、たくさんできるのですが、以下の3ファイルがとくに重要!というか、後はあまり見ません。
- accepted_hits.bam:BAM フォーマットのアライメントファイル
- junctions.bed:Exon Junction情報をまとめたファイルで、UCSC BED track フォーマット。ジャンクションは2つのBEDブロックから成り、"maximal overhang" で指定した数以上の塩基によって構成しているはず。スコアはアライメントの数を示す。UCSC ゲノムブラウザに表示できる
- insertions.bed / deletions.bed:insertions/deletions情報をまとめたUCSC BED tracksファイル。
Insertions.bed の chromLeft は、Insertion直前の塩基を表し、
Deletions.bed の chromLeft は、Deletionする最初の塩基を表している、らしい
つづく
登録:
コメント (Atom)