日本のニュースでも話題になりましたが、これは最初、新種の大腸菌ではないかと報道されました。
(読売新聞6月3日「欧州で広がるO104、新種の可能性…WHO」)
やがて、ドイツと中国、その他のチームによってゲノムが読まれ、これが新種ではないものの、ハイブリッドな特徴を持つ厄介な菌であることがわかりました。 BGIによると、
このE.coliは、下痢原性大腸菌の一つのカテゴリーである腸管凝集性大腸菌 enteroaggregative E. coli (EAEC) の系統ですが、志賀毒素を産生するファージゲノムを自身のバクテリアゲノムに組み込み、さらに多薬剤耐性遺伝子をもゲノムに組み込んでいたことがわかりました。
環状ゲノムのサイズは 5,278 kbp で、ほかに 88 kbp, 75 kbp, 1.5 kbp の3つのPlasmidから成るとのこと。
通常、志賀毒素を出す遺伝子、多剤耐性遺伝子は、ファージが関係するのですが、このE.coliは自身のゲノムにこれら遺伝子を組み込んでいることから、暫定的にShiga toxin-producing enteroaggregative Escherichia coli (STpEAEC) と呼ばれているそうです。
腸管出血性大腸菌 enterohaemorrhagic E. coli (EHEC) に特徴は似ているけれど別の名前で呼ばれているんですね。知りませんでした。
さて、前置きが長くなりました。
このドラフトシーケンシングに用いられた機器が、半導体シーケンサー Ion Torrent PGM です。
(実はBGIは、PGMのほかにIlluminaのマシンも使って読んでいるのですがそれはさておき)
Ion Torrent社のホームページへ行くと大きく宣伝しているのがわかるでしょう。
今はまだ数少ない、PGMのデータ(この大腸菌のリード)はここから入手できます。
Sffフォーマット: http://lifetech-it.hosted.jivesoftware.com/docs/DOC-1621
Fastqフォーマット: http://lifetech-it.hosted.jivesoftware.com/docs/DOC-1516
もしかするとユーザー登録が必要かもしれません。
また、アプリケーションノートやビデオなどは、Ion Torrentを良く知るために大変役に立ちますのでお勧めです。
念のため申しますと、私は Ion Torrent 社及びライフテクノロジーズ社と、特別な関係、があるわけではありませんのでご安心下さい。
ひとつ断わりをいれておきますが、Ion Torrent社のサイトから落とせるデータは、BGIが読んだデータではありません。
ストレインが違います。
現在5つのストレインでゲノムが読まれていて
- TY2482 (BGI in collaboration with University Medical Centre Hamburg-Eppendorf)
- LB226692 (Life Tech in-house in collaboration with University of Muenster)
- H112180280 (Health Protection Agency, Colindale, UK)
- 2 isolates, unnamed (Gottingen Genomics Lab, Germany)
BGIはTY2482を読んでいます。
Ion Torrentのサイトから落とせるデータは、LB226692のデータです。
BGIのデータを使いたい方は、
NCBI SRA からは、SRX067313 で検索すると出てきます。
BGIのサイトからは、ftp://ftp.genomics.org.cn/pub/Ecoli_TY-2482フォーマットはfastqです。
解析、de novo assembly については、彼ら(BGI)のワークフローが参考になるかと思います。
バクテリアゲノムアセンブリを初めてされる方は見ておいて損は無いでしょう。
http://climb.genomics.cn/Ecoli_TY-2482
見てわかる通り、BGIのチームは、IlluminaとIon Torrentの両方で読んで、アセンブルしました。
Ion Torrent PGM データのアセンブリに、Newbler を使ったのですね。
Roche 454 と同じ sff フォーマットなのでこれが使えるのでしょう。
PGMは、sff を出した後、fastqでも出力してくれます。 これならvelvetなどフリーツールでも使えますね。
現在、PGM に付属するソフトには、残念ながら独自のアセンブリツールが無いので、今のところPGMを使って de novo assembly をしようとすると、アセンブリソフトを別に求める必要があります。
さて、Ion Torrent のサイトから入手できる、LB226692のデータに話を戻しますね。
先のサイトから、sff のファイルをダウンロードしてきました。
8ファイル(8ラン分)あります。
私はRocheの機械を持っていませんし、アカデミアではなく企業の人間なので、タダでNewblerは使えません。
fastqを落としてアセンブってもいいがせっかく sff のフォーマットがあるのでできればsffのままアセンブりたい。
さて、、、どのアセンブリツールを使おうか。
手元にちょうどCLC Genomics Workbench があるのでそれでやってみることにします。
最初に言い訳がましくなりますが、このPGMのデータだけを使ってアセンブリしても、1本にはなりません。
BGIは、7ランのPGMのデータ、200x以上のIlluminaのシングルリード、さらにIlluminaペアエンド、を使って読んでやっとドラフトゲノムを完成させています。
私がこれからお見せしようとしているのは、PGMのsffデータだけでアセンブルしたら、どれくらいのContigができるのか、というものです。
CLC のGenomics Workbenchには、sff フォーマットがインポートでき、アセンブルも簡単にできます。
取り込んだ後の様子
アセンブルした後のマッピングした結果のContigリスト
Contigをクリックしたときの様子
データ量: 8ラン分のデータ、平均98bpの117万リード、合計114Mbp
アセンブルに要した時間: 1分10秒
マシンの性能: 64Bit Linux, メモリ24Mb
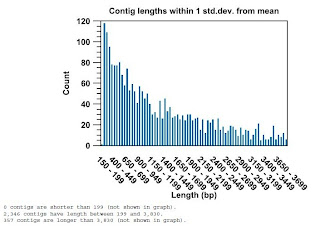
Contigについて(200bp以上の長さのみ)
2,700本のContigができました。 最長16,000 bpでした。
ふーん、こんなもんかな。
こんどはちゃんと配列の前処理をやってからランしようかと、思った次第です。
と、ここで終わってしまっては結構さみしいので、次に、BGIのTY2482のリードデータを使って、
Ion Torrent 7ラン分 + Illumina 200x分 のハイブリッドアセンブリをやってみました。
フォーマットはfastq です。 結果をまとめると、
データ量: 合計 20.5 M reads, 1.8 G bp
ランタイム: 15分
Contigについて
512本のContig、最長224,799 bp、N50 = 60,618 bp!
実はBGIはペアエンドでも読んでいるのですが、データは公開されていません。
ゆえにこれは、Ion Torrent PGM +Illumina GAII シングリリード のハイブリッドアセンブリの結果、です。
ショートリード(GAII)とロングリード(PGM)がうまく互いを補いながら、長くつないでいるのでしょうね。
ちなみにCLC Genomics Workbench のパラメータはデフォルトです。
このソフトについてはこちらを参照
http://www.w-fusion.com/J/CLC_wb.html
・・・・・・今回は少し長文になりました。
大腸菌についての参考資料
広島県保健環境センター研究報告,No.12,p1-12,2004
http://www.pref.hiroshima.lg.jp/hec/press/pdf/kenkyuhoukoku12/01.pdf










0 件のコメント:
コメントを投稿