というのはSanger型から始まってSolexa型、454型、SOLiD型、Ion Torrent型等、今までのシーケンサーの常識です。
でも、配列だけではなく、メチレーションの有無も同時に読んでくれたら・・・
これがPacBioではできる(らしい)のです。
Flusberg et al. Direct detection of DNA methylation during single-molecule, real-time sequencing.
Nat Methods. 2010 Jun;7(6):461-5.
昨年のthe Advances in Genome Biology and Technology (AGBT)のポスター(実際には行っていないんですけど)から注目していたのですが、PacBioのSingle Molecule Real Time (SMRT)シーケンスを使えば、塩基の修飾も配列と同時に検出することができるようなのです。
SMRTテクノロジーについては以前、
PacBio データの特徴 1 と 2 に書きました。
SMRTからの塩基配列データは、パルス (4種類の異なる光)として出力されます。
(AGBTポスターより)
パルスから得られる情報には、塩基そのものの他にも、一塩基が読みとられる時のパルスの幅 Pulse width (PW) と、次の塩基が読みとられるまでのパルスの間隔 Interpulse duration (IPD) もあります。
このIPDが、塩基が修飾されていると変化するのです。
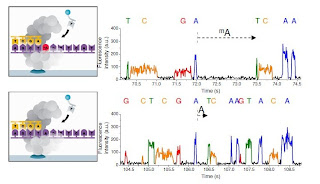
具体的には、Aがメチル化されている N6-methyladenosine (mA) の場合、Tと結合する部分のNにメチル化が起こっているため、Tと結合するのが遅くなるそうです。
そうすると、メチル化されたAのパルスの間隔 IPDが、メチル化されていないAと比べて大きくなります。 (下図の上がメチル化A、下がコントロールA。 メチル化Aの後、次のTまでの間隔が、コントロールのAからTまでの間隔と比べて大きいのがわかります)
この原理を用いて、メチル化されているアデノシン(に限らずシトシンも)を見つけよう! ということです。
でもこの検出方法は、うまくいくのでしょうか?
論文ではまず、人工的に作ったメチレーション配列とコントロール配列199塩基を、両方とも何百回も同じところを読んで、平均のメチル配列のIPDと、非メチル配列のIPDを比較しています。
これを見ると、必ずしもメチル化された箇所のIPDが大きく変化するわけではなく、その周辺の塩基のIPDがより影響を受けることがわかりました。
縦軸が塩基ごとのIPDの比(メチルサンプル/ 非メチルサンプル)の平均で、横軸が塩基のポジション、▲印がメチル化の場所です。
上からN6-methyladenosine、5-methylcytosine、5-hydroxymethylcytosine の順で、それぞれN=346、504、393 だそうです。
どうでしょう? メチル化された塩基の場所のIPDが変化しているものもありますが、その前後で大きく変化を示しているパターンのものもあるのがわかります。
N6-methyladenosineでは、メチル化塩基とその5番めの塩基
5-methylcytosineでは、2番、3番、6番めの塩基
5-hydroxymethylcytosineでは、2番、6番めの塩基
のIPDが大きく変化しています。 (縦軸の値が異なるのに注意)
このようなパターンがあることは、メチル化の種類も見分けることができる、ということです。
既存のバイサルファイト法のような形では不可能でしょう。
もちろん何百回も読まなくても、数回、しかし繰り返し同じ所を読むことで、メチル塩基と非メチル塩基を区別できるそうです。
SMRTのCircular topology型DNAテンプレートなら可能なのです。
赤線がメチルAを含むテンプレート、青線がコントロールです。
5回読めばはっきりメチルテンプレートとコントロールの、メチルAのパルスが区別できることを示しています。
人工配列はもういい、という方のために、彼らはDNA adenosine methyltransferase positive (dam+)の大腸菌から取り出したC. elegans fosmid 配列をも読んでいます。
比較対象は、Whole Genome Amplification(WGA)したコントロール配列です。
これは3.7kbの配列にフォーカスして解析しています。
dam+ のIPDをコントロール(WGA)のIPDで割った、IPD Ratio が縦軸です。
Aメチル化はGATC配列に起こりやすいので、non-GATCの塩基(○)とGATC塩基(■)を分けています。
N=106で、平均IPDをプロットしています。
全体的に、non-GATC塩基はIPD ratio が2以下なのに対し、GATC塩基(メチル化塩基)は、IPD Ratioが3から7の間にいるので、区別できる! ということは言えるかな?
ちょっと2500塩基付近の下のGATCが微妙ですが。
実際、これからの技術ですね。
IPDの変化のパターンは、メチル化の種類にだけ依存しているのか、周りの塩基によっても違うのか、生物種によっても違うのか、光学的な条件によっても違ってくるのか。
でも、希望は持てます。 一度パターンがわかってしまえば、シーケンスするだけでメチル化の種類もわかってしまうのですから。




0 件のコメント:
コメントを投稿