そのデータとは、E.coli O104-H4 の de novo sequence です。
このことを書いた後、ある方と、クオリティの話をしました。
ということで、本日はクオリティチェックの結果を書きます。
使ったファイルは、Ion Torrent 社のサイトから取得した、LB226692株のfastq ファイルの、64.fastq ファイルです。 本当は64.fastq - 71.fastq まで、8ファイル全部やりましたが、ここに示すのは64.fastq ファイルのみということで。
使ったツールは、FastQC と、PRINSEQ です。
両方とも同じようなツールです。
FastQCはPCの上で動くのに対し、PRINSEQはクラウド上で動くのが違いです。
ちなみにPRINSEQは、Internet Explore 9 ではまだうまく動かないみたいです。
FireFox、Google ChromeではOKでした。
FastQCの方が有名?
PRINSEQもいいですよ。
左下のUpload Fileをクリックして、ファイルを指定して、Upします。
でも難点は1つずつしかファイルをUPできないこと。
ではここから、
- リード長のばらつき
- リードにおけるクオリティの変化
- GCコンテンツ
リード長のばらつき (Length Distribution)
FastQC
PRINSEQ
108塩基長のリードが最も多いようです。
ばらつき具合はグラフから一目了然ですね。
リードのクオリティ (Read Quality Distribution)
FastQC
PRINSEQ
多くのシーケンサーデータ同様、Readの最初はクオリティが高いですね。
後ろの方に行くに従ってだんだんと低くなり、90塩基付近では10を切ってしまいます。
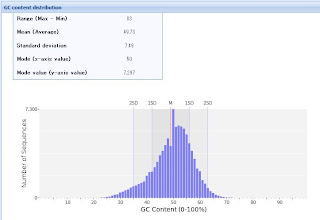
GC含量 (GC Content Distribution)
FastQC
PRINSEQ
確かE.coliのゲノムはGCリッチで50%位だったと思います。まちがってたらすみません。
このデータもリードのCG含量が平均50%ですので、まあ、想定内でしょう。
FastQCとPRINSEQを例に、リードのクオリティチェックをしました。
本当はもっとメニューがあります。
FastQCはWindowsでも動きます。
PRINSEQはインターネットにデータを送り、結果はWebブラウザで閲覧します。
とっても簡単なので、是非一度、自身でお試しください。







0 件のコメント:
コメントを投稿